谷劲松
(济南大学生物科学与技术学院)
摘要:冰岛温泉微生物是适合在极端环境条件下生存的微生物菌群聚集体,为人们研究微生物菌群耐受性、古生菌种类以及 CRISPR-Cas系统等作出了巨大贡献。冰岛温泉微生物体系的建立与完善对不可培养微生物和微生物基因库的全面深入研究起至关重要的作用。本文介绍了冰岛温泉微生物中的物种资源、分离方法及遗传多样性,重点论述了冰岛硫化叶菌CRISPR-Cas系统作为高效的基因编辑工具的发展现状,最后总结了冰岛温泉微生物的研究价值,并对其未来发展方向进行了展望。
关键词:冰岛温泉微生物;微生物多样性;古生菌;CRISPR-Cas系统
Research Progress of Microbe in Iceland Hot Springs
Gu Jinsong
Abstract: Iceland's hot spring microorganisms are suitable for living in extreme environmental conditions. They have made great contributions to the study of the tolerance of microflora, the species of Palaeozoic bacteria and the CRISPR-Cas system, etc. The establishment and perfection of Iceland Spa microbial system plays an important role in the study of non-culturable microorganisms and microbial gene pool in an all-round and in-depth way. The species resources, isolation methods and genetic diversity of microbe in Iceland hot spring were introduced in this paper. The development status of CRISPR-Cas system as an efficient gene editing tool was emphatically discussed. Finally, the research value of microbe in Iceland hot spring is summarized, and the future development direction is prospected.
Keywords: Iceland Spa Microorganism; Microbial Diversity; Palaeozoic; CRISPR-Cas System
一、引言
冰岛(Iceland)地理位置处于欧洲的最西部,靠近于北极圈。虽然隶属于欧洲国家,但离欧洲大陆比较远,离北美洲更接近。冰岛海岸线约为4970公里,领海的宽度为12海里,其大陆架面积达到11.1万平方公里[1-2]。因为这种优越的地理环境也造就了冰岛拥有了丰富的地热资源,温泉的数量列居世界首位,全岛约有250个碱性温泉-数量居世界首位,与此同时,岛上的火山数量也很多,所以被著称为“极圈火岛”,也被称为“冰火之国”。温泉的种类有很多:根据其活动的形态,可以分为涌出泉、喷泉、间缺泉等;其按照成分分为硫泉和碳酸泉[3]。高温硫泉区位于高温地热区,最大的位于托尔法冰原地区和格里姆斯弗顿地区。硫泉中含有的多种矿物质将这些温泉的周围“染色”[4]。区分温泉和蒸汽喷泉其实不容易,那是因为部分温泉也大量喷出蒸汽。蒸汽喷泉大多都处于活火山带,岩浆侵入地表附近,加热地下水成为酸性的蒸汽,而蒸汽孔被称为硫气孔[5]。如果地表水和地下水流入到硫气的孔中时间,那蒸汽喷泉周围的泥土会变成青黑和灰色的泥浆沸腾,就像锅中煮沸的粥一样沸腾翻滚,因此文人称它为“恶魔的厨房”。冰岛温泉中含有大量矿物质,每升温泉水所含矿物质可以达到1000 mg,其中主要成分有硅酸、氯化钠、硫酸钠、碳酸钠、钙、镁、氟和硼[6]。冰岛温泉拥有独特的理化特征,促进了居住在适合极端环境条件下特殊微生物群体的形成。与此同时,冰岛温泉提供了一个独特的环境,用于研究微生物群落对地球化学的响应,也正是因为这些环境中蕴藏着的多种微生物,它们参与氢气(H2)、硫(S)、铁(Fe)和砷(AS)的各种代谢过程,并具有高温和广泛的pH值[7]。同时,通过研究冰岛微生物群体中细菌培养与群落组成分子样本不一致的基础,开始培养出16S rRNA序列易于检测的菌株。通过将所检测到的遗传多样性与温泉垫系统典型的自然环境梯度相结合,阐明了基因与物种多样性之间的关系,揭示了物种发生的生态模式。
二、冰岛温泉微生物多样性
极端微生物已知在各种极端条件下生存,如高温或低温、高盐度、酸性和碱性pH值和高辐射[8]。而这些极端微生物在高温的环境中,如温泉,通过能在这种条件下保持催化活性的酶的帮助,超级嗜热菌才有了能力生存。同事从温泉中分离出的许多新型嗜热菌的详细基因组分析揭示了它们在工业和生物工艺中的潜在应用,通过使用元组学和下一代测序技术,可以发现这类在很大程度上不可培养的嗜极生物群落的惊人的基因组的多样性和复杂性,并且还发现了许多新型的嗜热细菌和病毒高质量基因组,其都具有若干潜在的应用前景。嗜热等适合极端环境条件生存的微生物就聚集于温泉环境,而其温泉在自然流放的过程中会因为有微生物包括原生动物、藻类、真菌以及细菌的存在,温泉池也可能因为人类活动而导致致病性微生物的污染[9]。
温泉微生物多样性的影响因素还包括温泉所处的地理位置,温泉的温度和酸碱度,硫、硼、铁、砷等水化学成分及氧气、光照等生态因子等。这些因素独立或协同影响温泉微生物的多样性[10]。从冰岛来看,地理位置不同的各个温泉其细菌和古菌的群落组成就会不同,虽然也有部分克隆与地理位置遥远的温泉的某些克隆在基因序列上有相似度很高的部分,但总体群落是不同的构成。例如:嗜酸菌分布在酸性矿物质水、酸性热泉等地区,再比如氧化硫杆菌在pH值低于0.5的环境中仍能存活,专性自养嗜酸的氧化亚铁硫杆菌(Thiobacillus ferrooxidans)能氧化硫和铁,并产生硫酸[11]。
嗜热微生物及其酶分解生物质的能力因其快速的反应时间、热稳定性和能降低污染风险的特性而受到人们的关注。嗜热微生物(最适生长温度50°C以上)是嗜热酶(如淀粉酶、纤维素酶、几丁质酶、果胶酶、木聚糖酶、蛋白酶、脂肪酶和DNA聚合酶)的来源,具有独特的理化性质,适合在高温下进行生物加工。此外,它们在许多溶剂、洗涤剂、酸性和碱性pH值条件下更稳定。其中最重要的酶是蛋白酶:碱性蛋白酶在洗涤剂中具有很大的稳定性,蛋白酶在洗衣粉、食品工业、皮革加工、制药等生物工业和生物学研究中有着广泛的应用。此外,纤维素酶在从农业纤维素原料中生产葡萄糖原料以及从可再生农业残留物中生产生物乙醇和附加值有机化合物方面具有巨大的商业潜力[12]。各种酶在生物工业中有着重要的应用;例如,蛋白酶和淀粉酶一起应用于食品工业、洗涤剂工业和制药等许多行业。另一种重要的酶明胶酶作为药物开发的靶点,是由于其在与肿瘤转移相关的结缔组织降解中起着重要作用。开发高效的耐热糖苷水解酶(GHS)可以加速生物燃料和生物化工的工业化。然而,对高温下生物质降解十分重要的嗜热菌及其酶的全谱还没有得到彻底的研究。由于嗜热微生物是针对生长温度最适宜为45℃以上的微生物,所以在冰岛微生物的种类中,嗜热微生物不仅对高温耐受力强,而且能够在高温下生长繁殖,因此生存需要高温度的环境。并且它与一般的芽孢细菌的耐热性不同,普通芽孢细菌在高温下形成芽孢抵抗逆境,待环境条件恢复,芽孢萌发成营养体,而芽孢不具有繁殖能力,只是抗逆性休眠体[13]。与此之外,嗜热细菌对高温环境的的另一个武器是被称为热休克蛋白的特殊分子,它们可以充当分子的保护者防止粘附的蛋白质的分散,在热环境中与分散的蛋白质可以积极地粘合在一起,再使其发挥功能。而且,与正常细菌相比,嗜热细菌具有高度密度和紧凑性的蛋白质,从而保护其免受降解[14]。此外,每种蛋白质复杂的3D结构之间的不同部分可能具有的额外的联系以帮助维持分子结构的稳定。这些策略使得微生物能够在我们认为的极端环境中存活并不断超越极限。科学家们认为,嗜热微生物具有特殊的生物机制,让聚合物(蛋白质和核酸)在高温下维持稳定的结构和功能。嗜热细菌的膜也不同,这种细菌膜富含更多饱和脂肪酸[15]。因为古生菌的膜异常结实,有异甲基醚这样的特殊脂肪,这种情况也是可能的。
生态模式允许我们假设细菌多样性,就像植物和动物之间的多样性一样,很可能是由进化辐射产生的,至少在一定程度上是由进化辐射引起的,这些辐射导致了生态上不同的物种。与森林群落一样,这种“微生物林”中的不同物种分布在环境梯度(例如温度而不是湿度或海拔)上,在给定的地点,适应遮荫的物种可能生活在林冠物种之下[16]。在冰岛微生物的研究中将通过在微观环境尺度上绘制物种相对于环境参数的分布图,进一步了解如何有选择地培育我们检测到的各种物种。因此,能够获得更多关于地球上所有生命所依赖的自然微生物群落错综复杂的预测知识。由于与应用问题(包括疾病)有关的微生物也必须遵守决定它们存在、分布和适应度相关的进化和生态规则,但也从中更好地挖掘到尚未在众多未培植微生物中发现的巨大生物技术潜力。
三、冰岛温泉古生菌
对于冰岛温泉来说,微生物物种中细菌种类比较多,主要分布在蓝藻门、变形菌门、拟杆菌门、硬壁菌门、放线菌门、绿藻门、产水菌门、绿弯菌门、异常球菌-栖热菌门等,优势菌群多为蓝藻门、变形菌门和产水菌门。古菌主要是广古菌和泉古菌[17]。
截至目前的研究发现,古菌在地球上多样的生态系统中广泛分布,例如极地海水、热泉、海底热液口、盐湖及海洋沉积物中;硫化叶菌(Sulfolobus)是最早发现的超嗜热嗜酸古菌,它属于TACK中的一个属,目前研究较多的有S.acfctocaldarius、S.colfatarius、S.islandius、和S.tokodaii四个种。它们好氧生长,最适生长温度75-80摄氏度,最适PH值2-3,可耐受PH为0.9-5.8,兼性自养型,既可以以二氧化碳为碳源,以硫代硫酸盐为能源营自养生活,也可以以蛋白胨、蔗糖或者酵母提取物作为碳源和能源异养生活[18]。
冰岛硫化叶菌(Sulfolobus islandicus)是一种最早分离自冰岛热泉中的极端嗜热古菌。其生存温度在70-85℃,pH2-3.5范围的矿物热泉中,除了冰岛,还在美国、俄罗斯等国家和地区的地热温泉中被发现,是一类广泛分布于北半球的嗜热古菌[19]。冰岛硫化叶菌是硫化叶菌家族中的一大类,由于其分布广泛,目前已发现21个不同的亚型,多数的基因组测序已全部完成。同时,由于硫化叶菌的培养条件易于实现,该菌种也就成为北半球最主要的能够培养的硫化叶菌之一。冰岛硫化叶菌(Sulfolobus islandicus)是古菌界泉古菌门的一种嗜酸嗜热微生物,因其易于培养且富含病毒和质粒等遗传因子而受到科学大众的广泛研究。
古菌很多基本的特征相近的是,在硫化叶菌中,参与能量代谢的蛋白质大多与细菌相似,而在DNA复制、修复、重组、细胞周期调控、转录、参与翻译等的蛋白质也是与真核生物具有同源性。分离叶菌具有类似的功能组分,而相比于真核生物来说更加简单[20],其蛋白耐热性的特点是针对特别有利的核异源性表达的纯化和结构进行分析,加之硫化叶菌能非常方便的在实验室进行培养[21],且其中含有丰富的染色体外遗传因子,硫化叶菌也就成为的古菌生物化学和遗传研究的模型生物[22]。
四、冰岛温泉微生物分离和遗传多样性
传统微生物的研究是采用分离培养的方法,温泉微生物的最佳生长条件与一般的微生物非常不同,因此,温泉微生物的研究受到限制[23]。在过去的二十多年里面,分子生物学技术,特别是16S基因技术,广泛用于鉴定未知细菌的研究,包括PCR-RFLP、PCR-RAPD、FISH、PCR-DGGE/TGGE、基因芯片等[24],确立了基于分子系分析的现代微生物分子的研究方法。近年来,伴随着高通量序测定技术的发展和宏观基因组学的方法的出现,温泉微生物群的多样性和遗产基因多态性研究备受关注[25]。以454焦磷酸测定为代表的高通量测定技术,可以更彻底地覆盖环境微生物群,发现稀少种类的温泉微生物,也能更详细地研究和理解温泉微生物的多样性和遗传基因的多样性。这种排序方法的基本原理是利用焦磷酸盐分析生物发光DNA序列的新技术。通过DNA聚合酶、ATP硫氧化酶、荧光酶和双磷酸酶的协同作用,将所有引物上每一个dNTP的聚合与一次荧光信号释放偶联起来。通过检测发出荧光信号的有无和强度,能够实时测定DNA序列。该技术不需要荧光标记的引管或核酸探针,也不需要电泳。分析结果具有快速、准确、灵敏、自动化的特点。而且,温泉微生物的多样性研究有助于认识高温环境中微生物遗传多样性和功能的多样性。通过明确在温泉中嗜热菌的多样性和与环境的关系,对认识生命的起源和进化有意义[26]。
温泉微生物遗传多样性是指温泉微生物在基因水平上所携带的各类遗传信息和遗传物质的总和。不同种群微生物之间的遗传物质和基因表达是具有很大的差异[27]。除了组成核酸分子碱基数量的巨大性和排列顺序的多样性外,在DNA复制中,其出现的碱基对变化、双链/单链DNA、双链/单链RNA等多种遗传形式的存在,转导、转化和接合等微生物特有的基因重组现象,使微生物遗传多样性大大扩展[28]。该水平上微生物多样性的研究要求高质量地获得生物大分子,对生物大分子进行基因层面的操作并进行精细的程序化检测[29]。大部分研究采用16S rRNA基因或16S-23S rRNA ITS (转录间隔区序列)分析法比较冰岛温泉微生物之间的基因差异性。从目前的研究来看,以16S rRNA作为分子标记的应用最为广泛。16S rRNA基因是细菌上编码rRNA相对应的DNA序列,其存在于所有细菌的基因组中。16S rRNA具有高度的保守性和特异性以及该基因序列足够长(包含约50个功能域)。随着PCR技术的出现及核酸研究技术的不断完善,16S rRNA基因检测技术已成为病原菌检测和鉴定的一种强有力工具。数据库的不断完善,应用该技术可以实现对病原菌进行快速、微量、准确简便地分类鉴定和检测。该技术主要有三个步骤:首先是基因组DNA的获得,其次是16S rRNA基因片段的获得,最后是进行16S rRNA基因序列的分析[30]。
而上文刚刚介绍的硫化叶菌最先发现和分离的株系来自冰岛的热泉[31],随后人们对冰岛硫化叶菌多个株系进行了全基因组测序,并鉴定到很多病毒和质粒等遗传元件。研究人员在此基础上成功构建了适用于冰岛硫化叶菌的载体以及高效的转化方法,实现了多种策略的基因敲除。并随着近年来冰岛硫化叶菌CRISPR-Cas系统被开发成一种高效的基因编辑工具[34],使其遗传操作体系得到了补充和完善。精确和有效的基因组修改在试图理解特定基因和其他遗传元素在生物学过程中以及在各种疾病中的作用方面具有很大的价值。CRISPR系统中有一种特殊的核酸酶-Cas9,它起着关键性的作用,这是由于引入了CLIS-CAS(分簇定期间隔的短回文重复序列-CRISPR)系统。该系统建立在细菌和考古学机制的基础上,负责获得抗噬菌体感染和转移外来遗传物质的免疫。微生物将外源DNA片段整合到其基因组中的CRISPR位点中,从而能够快速识别和消除可能会发生的感染[32]。原核生物中有几种CRISPR-CA系统,但在基因组工程中只使用CRISPRⅡ型的元件。CRISPR-Cas II型利用小RNA分子(crRNA和tracrRNA)精确地引导效应核酸酶Cas9到基因组中的一个特定位点,即与crRNA互补的序列。Cas9可用于:(1)在基因组中引入稳定的变化,例如在产生敲除和敲入的动物和细胞系的过程中,(2)激活或沉默活化基因的表达,以及(3)活细胞基因组中特定位点的可视化。基于CRISPR-Cas的工具已经成功地用于产生一些疾病的动物和细胞模型,例如特定类型的癌症。未来,可编程核酸酶的基因组编辑将在医学上得到广泛的应用,如某些遗传疾病的治疗和HIV感染者的治疗。与此同时,CRISPR-Cas系统保护原核生物免受噬菌体和可移动遗传元素的侵害,并成为基因工程革命性工具的基础。第二类CRISPR-Cas系统使用单个Cas内切酶与导向RNA结合,切割互补核酸靶点,使可编程序列特异性靶向性能以最小的机制实现。
冰岛硫化叶菌中含有一个 I-A 型和两个 III-B 型 CRISPR-Cas 系统,迄今为止对于其核酸干涉机制的研究已经较为清楚。该方法是在基因沉默质粒 pSe-Rp 基础上用相同方法克隆具有 DNA 干涉活性的 Spacer到2个 Repeat 之间组成 mini-CRISPR,再将经 SOE得到的供体 DNA (Donor DNA)用 Sal I 和 Not I 两个酶切位点克隆到该质粒上,最终得到目的基因编辑质粒 pGE[33]。pGE 上 mini-CRISPR 转录的 crRNA 可以介导核酸 Cas9干涉复合物特异性切割目的基因,供体 DNA 上可以设计缺失、插入和点突变,用于与基因组上编辑位点发生同源重组从而使细胞存活。用于基因组编辑S. islandicusis的I-A型和III-B型CRYSPR-Cas系统重新利用的原理,islandicusis在由质粒携带的CRYSPR阵列产生的CRRNA引导自身靶向性的情况下,其通过靶向相应的原间隔物用于DNA破坏选择性地杀死野生型细胞,并选择性地保留突变细胞,同时因为突变染色体在相应的位置含有非靶序列,因此没有自我靶向性[34]。
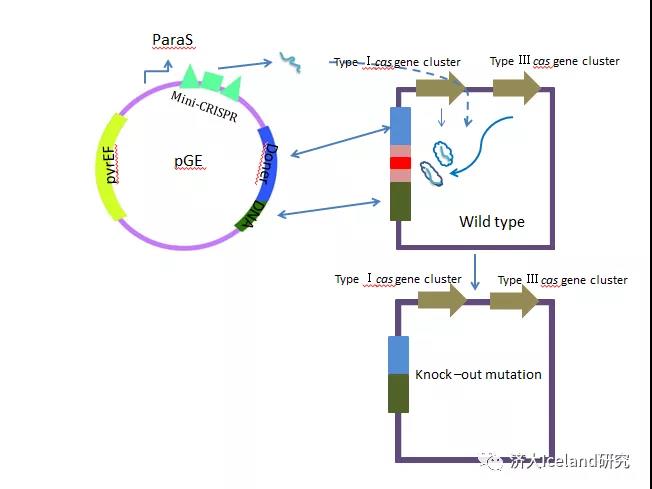
图1 基于CRISPR-Cas 系统的基因编辑法
Figure 1 Gene Editing method based on CRISPR-Cas system
我们得知了冰岛硫化叶菌遗传操作体系的基本元素,分析了现有基因编辑方法的优缺点,并在此基础上为选择合适的方法、对冰岛硫化叶菌进行遗传改造提供一定的指导[35]。对全球不同区域的硫化叶菌的基因序列比较研究究表明,硫化叶菌由于地理的分隔而具有了很明显的地域特征,所以,对硫化叶菌的深度研究也有助于我们进一步认识在空间尺度上的微生物基因组进化。除此以外,冰岛发现的硫化叶菌中也具有获得性免疫系统——CRISPR系统,这对于研究古菌与细菌的共同起源问题也具有一定的意义[36]。
五、结语
其中最重要的是,生活在温泉中的微生物能在可以在杀死大多数生物的环境下生存,这对理解地球生命起源和早期进化非常关键。热泉为我们提供了史前地球生命诞生和进化的线索,那时候地球上几乎没有阳光、光合作用或者植物。这意味着,稀薄的原始大气层中可能没有氧气。
对冰岛温泉微生物多样性的研究不仅是探索生命的重要手段,同时也为许多超常物质的研究开发提供了丰富的资源。生物技术在温泉微生物研究中的应用使许多热稳定酶合成相关基因的筛选和克隆成为现实。也通过测序等方法促进了温泉中不可培养微生物的发现和发展,丰富微生物基因库,带来巨大的经济效益促进了类似冰岛等地热资源丰富国家科学和旅游业的发展。
参考文献
[1] Keeling PJ, Klenk HP, Singh RK, et al. Sulfolobus islandicus plasmids pRN1 and pRN2 share distant but common evolutionary ancestry[J]. Extremophiles, 1998, 2(4): 391-393. DOI:10.1007/s007920050083.
[2] Purschke WG, Schäfer G. Independent replication of the plasmids pRN1 and pRN2 in the archaeon Sulfolobus islandicus[J]. FEMS Microbiology Letters, 2001, 200(1): 97-102. DOI:10.1111/fml.2001.200.issue-1.
[3] Lipps G. The replication protein of the Sulfolobus islandicus plasmid pRN1[J]. Biochemical Society Transactions, 2004, 32: 240-244. DOI:10.1042/bst0320240.
[4] Lipps G, Röther S, Hart C, et al. A novel type of replicative enzyme harbouring ATPase, primase and DNA polymerase activity[J]. The EMBO Journal, 2003, 22(10): 2516-2525. DOI:10.1093/emboj/cdg246.
[5] Lipps G, Stegert M, Krauss G. Thermostable and site-specific DNA binding of the gene product ORF56 from the Sulfolobus islandicus plasmid pRN1, a putative archael plasmid copy control protein[J]. Nucleic Acids Research, 2001, 29(4): 904-913. DOI:10.1093/nar/29.4.904.
[6] Lipps G, Ibanez P, Stroessenreuther T, et al. The protein ORF80 from the acidophilic and thermophilic archaeon Sulfolobus islandicus binds highly site-specifically to double-stranded DNA and represents a novel type of basic leucine zipper protein[J]. Nucleic Acids Research, 2001, 29(24): 4973-4982. DOI:10.1093/nar/29.24.4973.
[7] Berkner S, Grogan D, Albers SV, et al. Small multicopy, non-integrative shuttle vectors based on the plasmid pRN1 for Sulfolobus acidocaldarius and Sulfolobus solfataricus, model organisms of the (cren-)archaea[J]. Nucleic Acids Research, 2007, 35(12): e88. DOI:10.1093/nar/gkm449.
[8] Peng N, Xia Q, Chen ZJ, et al. An upstream activation element exerting differential transcriptional activation on an archaeal promoter[J]. Molecular Microbiology, 2009, 74(4): 928-939. DOI:10.1111/j.1365-2958.2009.06908.x.
[9] Woese CR, Kandler O, Wheelis ML. Towards a natural system of organisms: proposal for the domains Archaea, Bacteria, and Eucarya[J]. Proceedings of the National Academy of Sciences of the United States of America, 1990, 87(12): 4576-4579. DOI:10.1073/pnas.87.12.4576.
[10] Contursi P, Jensen S, Aucelli T, et al. Characterization of the Sulfolobus host-SSV2 virus interaction[J]. Extremophiles, 2006, 10(6): 615-627. DOI:10.1007/s00792-006-0017-2.
[11] Li YJ, Pan SF, Zhang Y, et al. Harnessing Type Ⅰ and Type Ⅲ CRISPR-Cas systems for genome editing[J]. Nucleic Acids Research, 2016, 44(4): e34. DOI:10.1093/nar/gkv1044.
[12] Cannio R, Contursi P, Rossi M, et al. An autonomously replicating transforming vector for Sulfolobus solfataricus[J]. Journal of Bacteriology, 1998, 180(12): 3237-3240.
[13] Thia-Toong TL, Roovers M, Durbecq V, et al. Genes of de novo pyrimidine biosynthesis from the hyperthermoacidophilic crenarchaeote Sulfolobus acidocaldarius: novel organization in a bipolar operon[J]. Journal of Bacteriology, 2002, 184(16): 4430-4441. DOI:10.1128/JB.184.16.4430-4441.2002.
[14] Deng L, Zhu HJ, Chen ZJ, et al. Unmarked gene deletion and host-vector system for the hyperthermophilic crenarchaeon Sulfolobus islandicus[J]. Extremophiles, 2009, 13(4): 735-746. DOI:10.1007/s00792-009-0254-2.
[15] Berkner S, Lipps G. Mutation and reversion frequencies of different Sulfolobus species and strains[J]. Extremophiles, 2008, 12(2): 263-270. DOI:10.1007/s00792-007-0125-7.
[16] Jonuscheit M, Martusewitsch E, Stedman KM, et al. A reporter gene system for the hyperthermophilic archaeon Sulfolobus solfataricus based on a selectable and integrative shuttle vector[J]. Molecular Microbiology, 2003, 48(5): 1241-1252.
[17] Grogan DW. Evidence that β-galactosidase of Sulfolobus solfataricus is only one of several activities of a thermostable β-D-glycosidase[J]. Applied and Environmental Microbiology, 1991, 57(6): 1644-1649.
[18] Peng WF, Feng MX, Feng X, et al. An archaeal CRISPR type Ⅲ-B system exhibiting distinctive RNA targeting features and mediating dual RNA and DNA interference[J]. Nucleic Acids Research, 2015, 43(1): 406-417. DOI:10.1093/nar/gku1302.
[19] Liu JF, Gao RX, Li CT, et al. Functional assignment of multiple ESCRT-Ⅲhomologs in cell division and budding in Sulfolobus islandicus[J]. Molecular Microbiology, 2017, 105(4): 540-553.
[20] Liu T, Liu ZZ, Ye Q, et al. Coupling transcriptional activation of CRISPR-Cas system and DNA repair genes by Csa3a in Sulfolobus islandicus[J]. Nucleic Acids Research, 2017, 45(15): 8978-8992. DOI:10.1093/nar/gkx612.
[21] Li YJ, Zhang Y, Lin JZ, et al. Cmr1 enables efficient RNA and DNA interference of a Ⅲ-B CRISPR-Cas system by binding to target RNA and crRNA[J]. Nucleic Acids Research, 2017, 45(19): 11305-11314.
[22] He F, Vestergaard G, Peng WF, et al. CRISPR-Cas type Ⅰ-A Cascade complex couples viral infection surveillance to host transcriptional regulation in the dependence of Csa3b[J]. Nucleic Acids Research, 2017, 45(4): 1902-1913.
[23] Han WY, Li YJ, Deng L, et al. A type Ⅲ-B CRISPR-Cas effector complex mediating massive target DNA destruction[J]. Nucleic Acids Research, 2017, 45(4): 1983-1993.
[24] León-Sobrino C, Kot WP, Garrett RA. Transcriptome changes in STSV2-infected Sulfolobus islandicus REY15A undergoing continuous CRISPR spacer acquisition[J]. Molecular Microbiology, 2016, 99(4): 719-728. DOI:10.1111/mmi.2016.99.issue-4.
[25] Liu T, Li YJ, Wang XD, et al. Transcriptional regulator-mediated activation of adaptation genes triggers CRISPR de novo spacer acquisition[J]. Nucleic Acids Research, 2015, 43(2): 1044-1055.
[26] Peng WF, Li H, Hallstrom S, et al. Genetic determinants of PAM-dependent DNA targeting and pre-crRNA processing in Sulfolobus islandicus[J]. RNA Biology, 2013, 10(5): 738-748. DOI:10.4161/rna.23798.
[27] Cao JJ, Wang Q, Liu T, et al. Insights into the post-translational modifications of archaeal Sis10b (Alba): lysine-16 is methylated, not acetylated, and this does not regulate transcription or growth[J]. Molecular Microbiology, 2018, 109(2): 192-208.
[28] Konermann S, Brigham MD, Trevino AE, et al. Genome-scale transcriptional activation by an engineered CRISPR-Cas9 complex[J]. Nature, 2015, 517(7536): 583-588.
[26] Peng N, Deng L, Mei YX, et al. A synthetic arabinose-inducible promoter confers high levels of recombinant protein expression in hyperthermophilic archaeon Sulfolobus islandicus[J]. Applied and Environmental Microbiology, 2012, 78(16): 5630-5637. DOI:10.1128/AEM.00855-12.
[27] Schleper C, Kubo K, Zillig W. The particle SSV1 from the extremely thermophilic archaeon Sulfolobus is a virus: demonstration of infectivity and of transfection with viral DNA[J]. Proceedings of the National Academy of Sciences of the United States of America, 1992, 89(16): 7645-7649. DOI:10.1073/pnas.89.16.7645.
[28] Albers SV, Driessen AJM. Conditions for gene disruption by homologous recombination of exogenous DNA into the Sulfolobus solfataricus genome[J]. Archaea, 2008, 2(3): 145-149.
[29] Aucelli T, Contursi P, Girfoglio M, et al. A spreadable, non-integrative and high copy number shuttle vector for Sulfolobus solfataricus based on the genetic element pSSVx from Sulfolobus islandicus[J]. Nucleic Acids Research, 2006, 34(17): e114. DOI:10.1093/nar/gkl615.
[30] Zhang CY, Tian B, Li SM, et al. Genetic manipulation in Sulfolobus islandicus and functional analysis of DNA repair genes[J]. Biochemical Society Transactions, 2013, 41(1): 405-410.
[31] Zhang CY, Guo L, Deng L, et al. Revealing the essentiality of multiple archaeal pcna genes using a mutant propagation assay based on an improved knockout method[J]. Microbiology, 2010, 156: 3386-3397. DOI:10.1099/mic.0.042523-0.
[32] Deveau H, Garneau JE, Moineau S. CRISPR/Cas system and its role in phage-bacteria interactions[J]. Annual Review of Microbiology, 2010, 64: 475-493. DOI:10.1146/annurev.micro.112408.134123.
[33] Mali P, Yang LH, Esvelt KM, et al. RNA-guided human genome engineering via Cas9[J]. Science, 2013, 339(6121): 823-826. DOI:10.1126/science.1232033.
[34] Cong L, Ran FA, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems[J]. Science, 2013, 339(6121): 819-823. DOI:10.1126/science.1231143.
[35] Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J]. Science, 2012, 337(6096): 816-821. DOI:10.1126/science.1225829.
[36] Gudbergsdottir S, Deng L, Chen ZJ, et al. Dynamic properties of the Sulfolobus CRISPR/Cas and CRISPR/Cmr systems when challenged with vector-borne viral and plasmid genes and protospacers[J]. Molecular Microbiology, 2011, 79(1): 35-49.